在前面的教程中,我们介绍了使用omicverse完成基本的RNA-seq的分析流程,在本节教程中,我们将介绍如何使用omicverse完成加权基因共表达网络分析WGCNA以及蛋白质相互作用PPI分析。
环境的下载
在这里我们只需要安装omicverse环境即可,有两个方法:
- 一个是使用conda:
conda install omicverse -c conda-forge - 另一个是使用pip:
pip install omicverse -i https://pypi.tuna.tsinghua.edu.cn/simple/。-i的意思是指定清华镜像源,在国内可能会下载地快一些。
导入环境
import omicverse as ov
ov.utils.ov_plot_set()
加权基因共表达网络分析(WGCNA)
加权基因共表达网络分析(WGCNA)是一种系统生物学方法,用于表征不同样品之间的基因关联模式,可用于鉴定高度协同的基因集,并基于基因集的内生性和基因集与表型之间的关联来鉴定候选生物标志物基因或治疗靶点。目前引用量已超过15,000。但Python中完成WGCNA分析相关的包仍是空白。我们根据WGCNA的原理,从底层上复现了原版WGCNA算法。
加载数据
在这里,我们选择WGCNA原版的演示数据来进行分析,数据可以在github上进行下载。
import pandas as pd
data=ov.utils.read_csv(filepath_or_buffer='https://raw.githubusercontent.com/Starlitnightly/ov/master/sample/LiverFemale3600.csv',index_col=0)
data.head()
相关性矩阵计算
WGCNA的第一步是计算基因间的相关性矩阵,这里我们采用皮尔森系数的计算方法,来完成基因间的直接相关性矩阵计算。
gene_wgcna=ov.bulk.pyWGCNA(data,save_path='result')
gene_wgcna.calculate_correlation_direct(method='pearson',save=False)
在 pyWGCNA 模块中,我们需要将直接相关矩阵转换为间接相关矩阵来计算软阈值,软阈值可以帮助我们将原来的相关网络转换为无尺度网络
gene_wgcna.calculate_correlation_indirect(save=False)
gene_wgcna.calculate_soft_threshold(save=False)
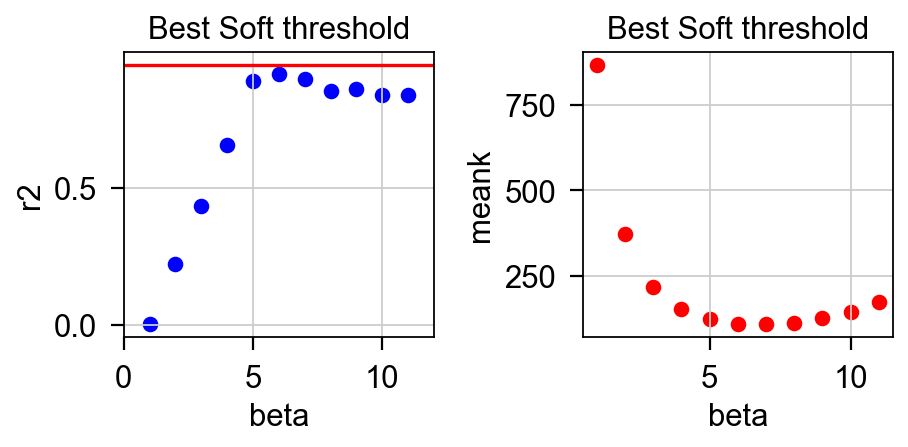
左边的垂直坐标是无尺度网络的评估指标r^2。R2越接近1,网络就越接近无尺度网络,通常需要r^2> 0.8或0.9。右侧垂直坐标为平均连通度,随 β 值的增加而减小。将这两个图结合起来,通常选择 r^2首次达到0.8或0.9或更高时的 β 值。利用 β 值,我们可以根据方程将相关矩阵转换成邻接矩阵。
然后我们构造拓扑重叠矩阵。
gene_wgcna.calculate_corr_matrix()
共表达网络分析
在获得基因间的拓扑重叠矩阵后,我们使用动态剪切树的方式来寻找基因间的模块。在这里,我们使用WGCNA作者发表的DynamicTree的算法来实现此功能。
gene_wgcna.calculate_distance()
gene_wgcna.calculate_geneTree()
gene_wgcna.calculate_dynamicMods()
module=gene_wgcna.calculate_gene_module()
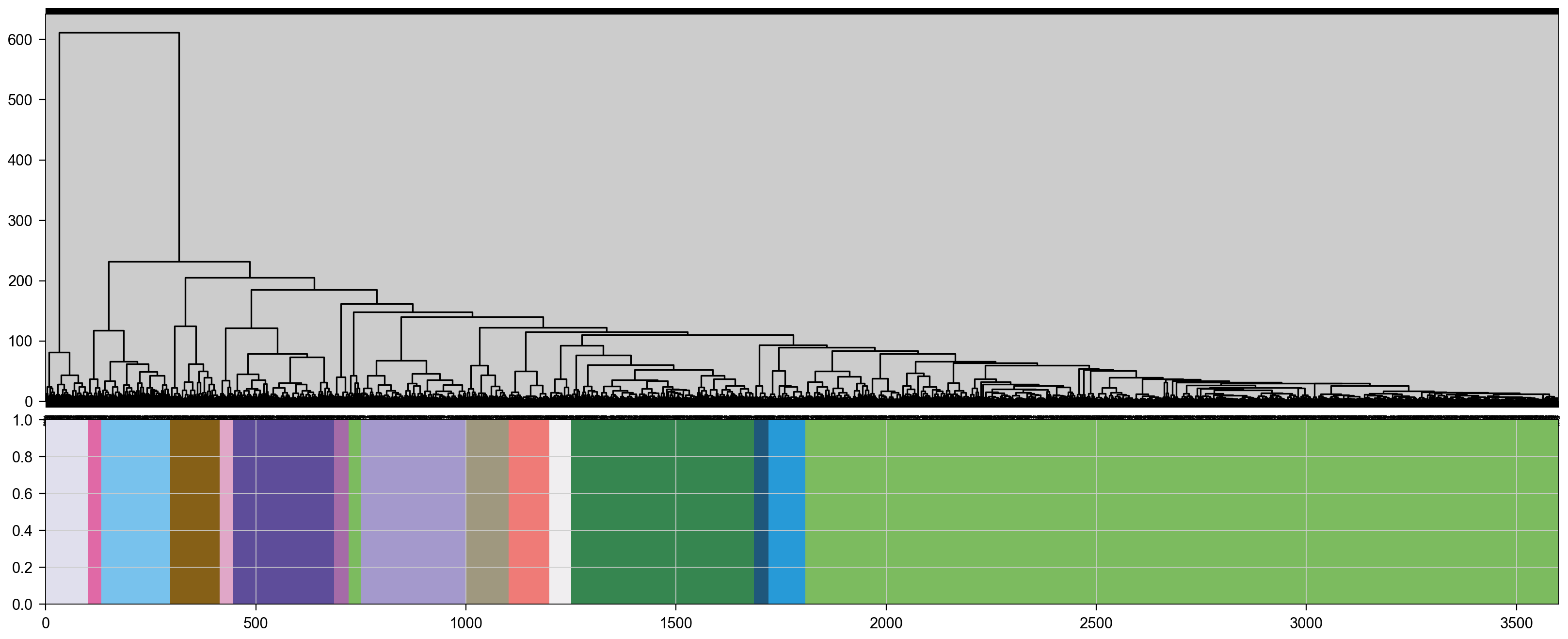
在这里,我们成功地计算了每个基因的模块,共有15个模块,它们的颜色都显示在图中。我们使用.head()查看每个基因所属的模块。
module.head()
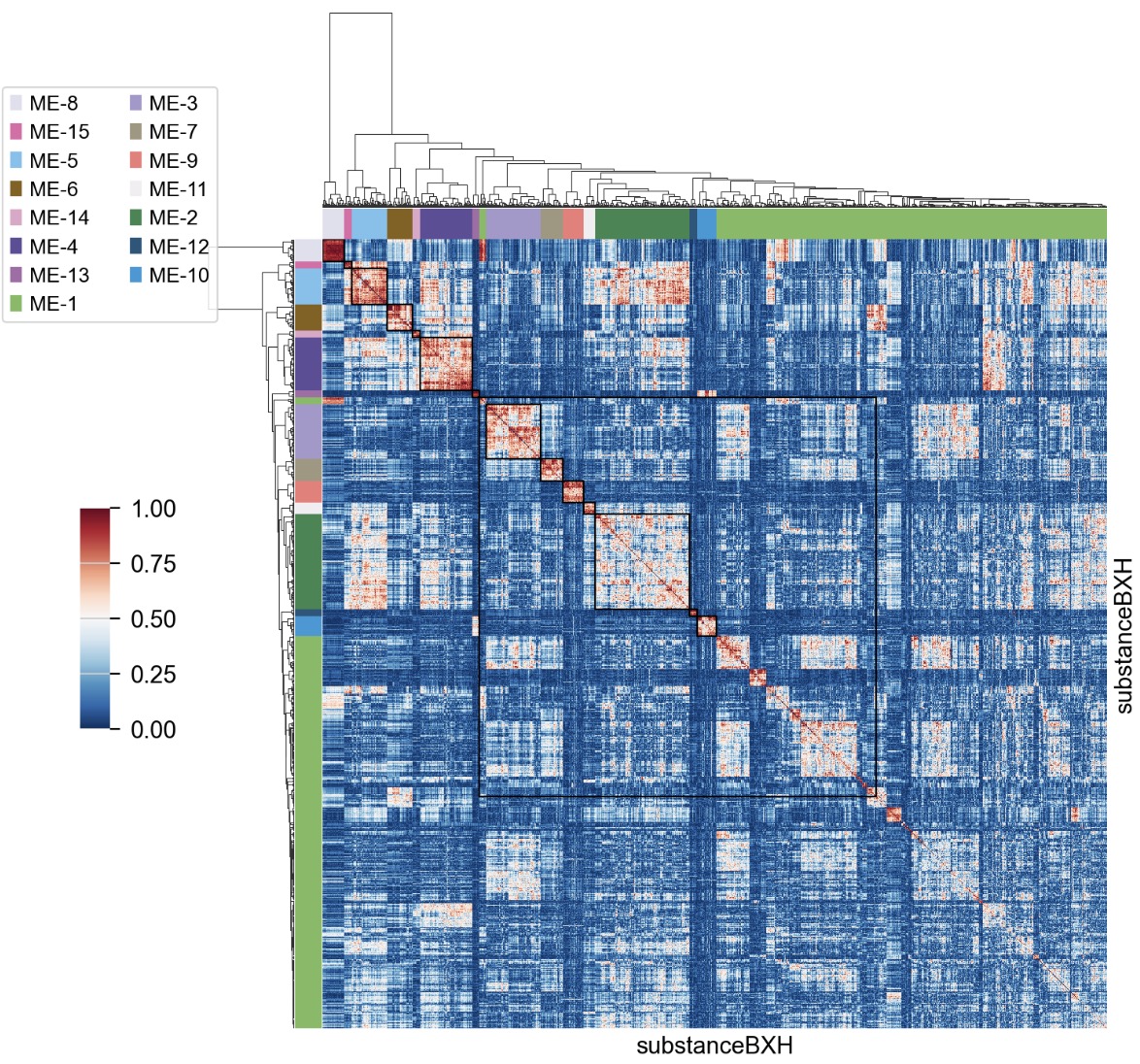
我们还可以使用.plot_matrix()来可视化拓扑重叠矩阵与模块之间的关系。
gene_wgcna.plot_matrix()
子模块分析
有时候我们对一个基因或一个通路的模块感兴趣,我们需要提取基因的子模块进行分析和定位。例如,我们选择了两个模块6和12作为分析的子模块
gene_wgcna.get_sub_module([6,12]).shape
我们发现模块6和模块12共有151个基因。接下来,我们使用之前构建的无尺度网络,将阈值设置为0.95,为模块6和模块12构建一个基因相关网络图。
sub_G=gene_wgcna.get_sub_network([6,12],correlation_threshold=0.95)
pyWGCNA 提供了一个简单的可视化函数 plot_sub_network 来可视化我们感兴趣的基因相关性网络。
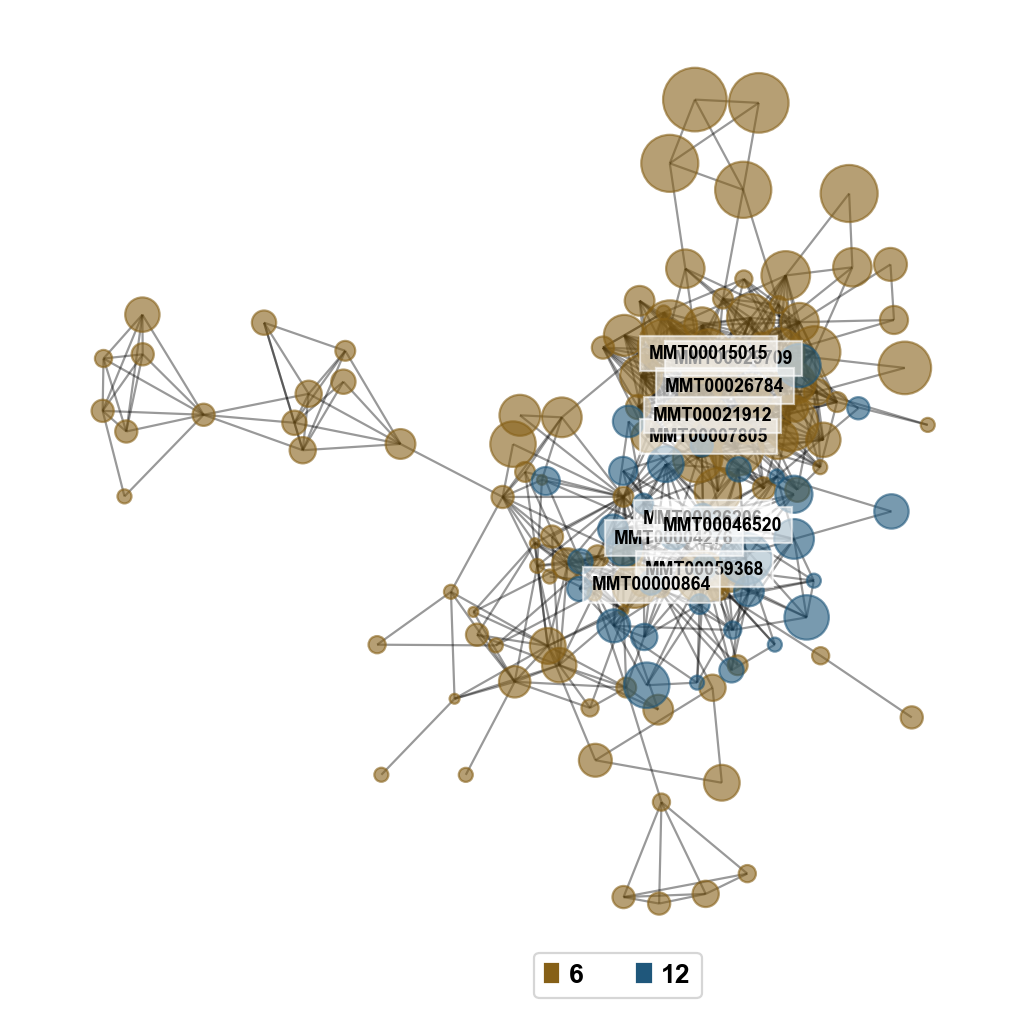
模块和性状相关性分析
除了可以根据目标基因选择模块外,我们还可以根据特定的样本性状选择模块。我们可以计算每个样本的性状和模块之间的相关性,从而找到与我们感兴趣的性状模块。
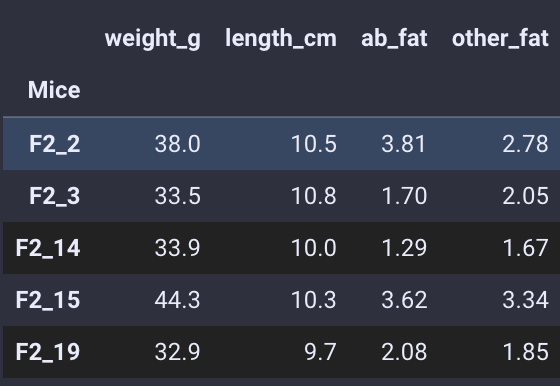
我们首先从 github 中读取特征矩阵。特征矩阵形状必须是以样本为索引,列为特征。示例名称必须与前面的原始数据的示例名称一致。
meta=ov.utils.read_csv(filepath_or_buffer='https://raw.githubusercontent.com/Starlitnightly/ov/master/sample/character.csv',index_col=0)
meta.head()
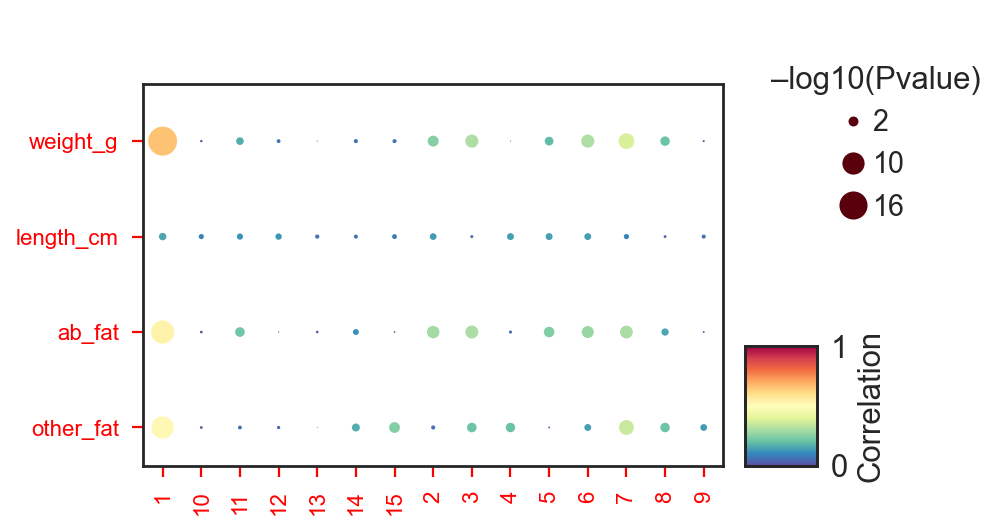
然后利用分析主成分分析和相关性,计算模块与性状之间的关系。
cor_matrix=gene_wgcna.analysis_meta_correlation(meta)
ax=gene_wgcna.plot_meta_correlation(cor_matrix)
蛋白质互作网络分析
我们接下来介绍蛋白质互作网络的分析,STRING 是一个已知和预测的蛋白质-蛋白质相互作用的数据库。这种相互作用包括直接的(物理的)和间接的(功能的)关联; 它们来源于计算预测,来源于生物体之间的知识转移,以及来自其他(主要的)数据库的相互作用。
在这里,我们制作了一个教程,使用omicverse来构建蛋白质-蛋白质相互作用网络。
演示数据
这里我们使用 string-db 的示例数据来执行分析
酵母中的 FAA4是一种长链脂肪酰辅酶 A 合成酶,它与其他合成酶以及调节剂有关。
酵母的NCBI taxonomy Id: 4932
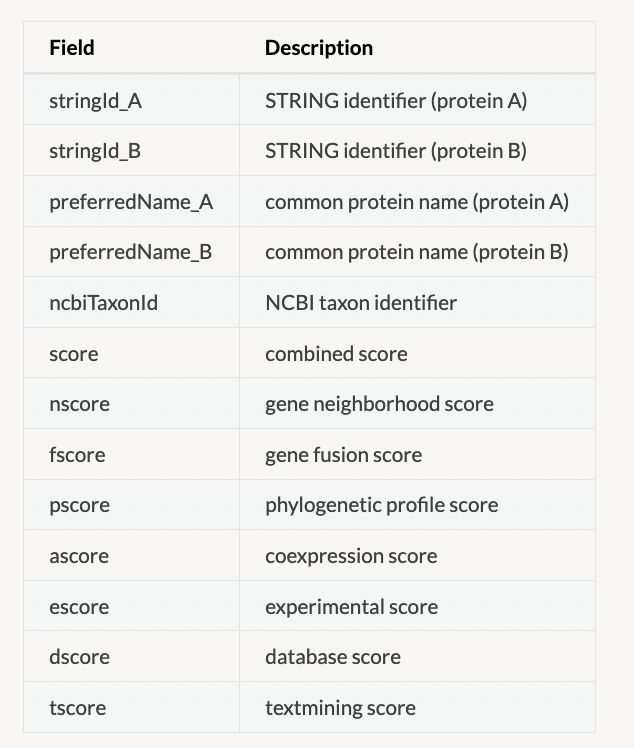
使用omicverse完成蛋白质相互作用网络分析需要三个数据:蛋白列表
,蛋白类别字典和蛋白颜色字典,颜色字典是绘图时的每个蛋白的颜色,一般与类别字典相同。在这里我们随机生成一个颜色字典和类别字典。
gene_list=['FAA4','POX1','FAT1','FAS2','FAS1','FAA1','OLE1','YJU3','TGL3','INA1','TGL5']
gene_type_dict=dict(zip(gene_list,['Type1']*5+['Type2']*6))
gene_color_dict=dict(zip(gene_list,['#F7828A']*5+['#9CCCA4']*6))
蛋白质相互作用关系预测
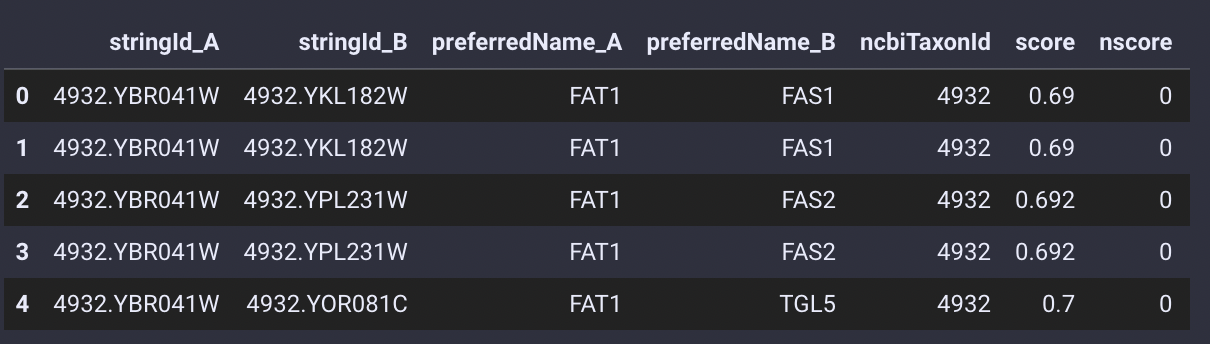
omicverse提供了一个十分简单的APIov.bulk.string_interaction,只需要输入蛋白列表即可完成相互作用关系的预测。
G_res=ov.bulk.string_interaction(gene_list,4932)
G_res.head()
蛋白质相互作用网络分析
当然,omicverse还有非常漂亮的可视化函数,来可视化蛋白质相互作用网络,在这里,我们需要使用pyPPI类来完成上述分析。
#4932代表酵母,人类一般是9606,小鼠一般是10090
ppi=ov.bulk.pyPPI(gene=gene_list,gene_type_dict=gene_type_dict,gene_color_dict=gene_color_dict,species=4932)然后我们连接到 string-db 来计算蛋白质-蛋白质的相互作用
ppi.interaction_analysis()
我们提供了.plot_network()来可视化蛋白质相互作用网络,更多的参数您可以使用help(ov.utils.plot_network)来寻找。
ppi.plot_network()
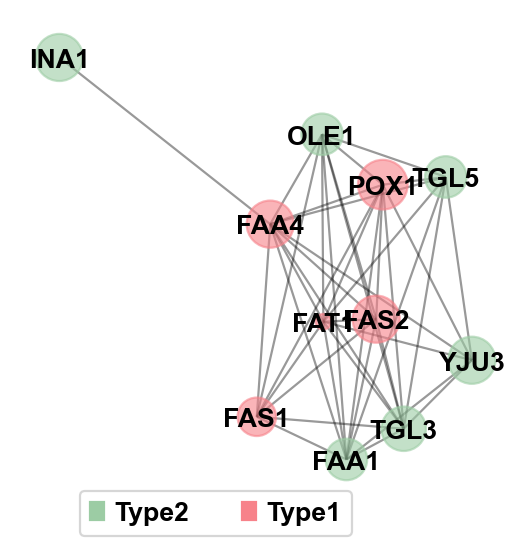
我们还可以使用ppi.G来获取蛋白质相互作用网络,该变量的格式为networkx,感兴趣的读者可以自行研究networkx包的相关分析。
到此,我们的教程就结束了,如果你认为本教程对你的研究有帮助,不要忘记引用omicverse和WGCNA,最后感谢Jimmy老师对omicverse的大力支持。